
Abstrak
Pencitraan spektrometri massa desorpsi/ionisasi laser berbantuan matriks (MALDI-MSI) adalah alat pencitraan baru yang memungkinkan visualisasi ratusan analit tanpa bias dalam satu eksperimen. Makalah ini menyoroti adaptasi MALDI-MSI dalam konteks berbeda pada tahun 2024, seperti diagnostik klinis, farmakologi, aplikasi forensik, metabolisme tanaman, dan biologi. Tantangan dan kemajuan juga dibahas terkait persiapan sampel, instrumentasi, analisis data, dan integrasi pembelajaran mesin dalam tren resolusi sel tunggal dan multiomik. Masih ada ruang untuk perbaikan dalam sensitivitas, resolusi spasial, algoritma akuisisi, dan integrasi data di seluruh data multiomik untuk mengaktifkan MALDI-MSI di tingkat subseluler.
1 Pendahuluan
Sejak pengembangan spektrometri massa (MS) pada awal abad ke-20, ia telah berkembang menjadi berbagai teknik khusus, termasuk pencitraan spektrometri massa (MSI), yang memungkinkan visualisasi distribusi molekuler di seluruh sampel biologis [ 1 ]. Dibandingkan dengan MS konvensional, seperti spektrometri massa tandem kromatografi cair (LC-MS/MS), yang menganalisis seluruh sampel dalam larutan sebagai entitas tunggal, MSI mengumpulkan spektrum massa dalam piksel virtual yang berjarak sama, dari 5 hingga 150 µm 2 , di seluruh bagian jaringan tipis. Data diperoleh sebagai spektrum individual per piksel, yang kemudian dihubungkan ke lokasi tertentu, yang memungkinkan pemetaan spasial analit. Teknologi ini memungkinkan beberapa analit dideteksi dalam eksperimen yang sama dalam pendekatan yang ditargetkan dan tidak ditargetkan. Oleh karena itu, ia telah muncul sebagai teknologi yang menjanjikan untuk studi penemuan yang bertujuan untuk menentukan lokalisasi analit dalam pendekatan yang tidak bias. Di sini, kami membahas tantangan dan kemajuan teknologi MALDI-MSI pada tahun 2024, dengan fokus pada persiapan sampel, instrumentasi, akuisisi data, dan analisis. Penerapan MALDI-MSI untuk berbagai aplikasi dan potensinya dalam multiomik spasial juga dijelaskan dalam tinjauan singkat ini.
1.1 Sejarah
MSI pertama kali dikonseptualisasikan lebih dari 50 tahun yang lalu, ketika MS dikombinasikan dengan spektrometri massa ion sekunder (SIMS), sebuah teknik analisis permukaan di mana ion-ion disemprotkan dari permukaan sampel dan kemudian dianalisis melalui MS [ 2 ]. Meskipun awalnya digunakan untuk menganalisis permukaan semikonduktor, MSI mulai mendapatkan perhatian luas di bidang biomedis ketika Caprioli [ 3 ] dan Spengler [ 4 ] menunjukkan penggunaan MALDI-MSI untuk analisis protein dan peptida. Teknik ionisasi lainnya juga dikembangkan, seperti desorption electrospray ionisation (DESI) dengan prosedur persiapan sampel yang kurang kompleks [ 5 ], dan laser ablation inductively coupled plasma (LA-ICP) untuk analisis unsur [ 6 ].
Terdapat peningkatan linear dalam studi MSI dari tahun 2010 hingga 2024 (Gambar 1A ). Sebagai teknik ionisasi lunak yang mencakup rentang massa yang luas, MALDI memungkinkan deteksi spektrum senyawa yang luas mulai dari metabolit kecil hingga protein besar [ 7 ], sehingga menjadi salah satu sumber ion yang paling umum digunakan (Gambar 1B ). Lintasan kemajuan terkini untuk meningkatkan sensitivitas, resolusi, dan throughput dalam MALDI-MSI, terutama pada tahun 2024, terus meluas ke lebih banyak aplikasi, yang akan menjadi pusat perhatian dalam tinjauan mini ini.
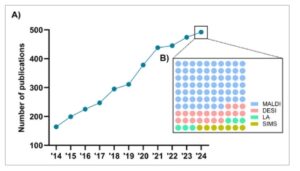
GAMBAR 1
Buka di penampil gambar
Tren publikasi studi MSI. (A) Grafik garis yang menunjukkan jumlah publikasi yang terdaftar di PubMed dengan kueri penelusuran ‘pencitraan spektrometri massa’ dari tahun 2014 hingga 2024. (B) Representasi proporsional dari empat sumber ionisasi yang disebutkan dalam studi MSI yang dipublikasikan pada tahun 2024. Kueri penelusuran yang digunakan adalah ‘pencitraan spektrometri massa’ DAN ‘MALDI’ ATAU ‘SIMS’ ATAU ‘DESI’ ATAU ‘ablasi laser’. Data diperoleh per 31 Desember 2024.
1.2 Prinsip Kerja MALDI-MSI
Gambaran umum alur kerja MALDI-MSI ditunjukkan pada Gambar 2. Alur kerja MALDI-MSI umum untuk analisis jaringan dimulai dengan pengawetan sampel. Bergantung pada analit yang dimaksudkan untuk deteksi, metode yang paling umum digunakan adalah fiksasi formalin untuk pemotongan mikrotom, atau pembekuan cepat menggunakan nitrogen cair untuk mengawetkan jaringan segar untuk pemotongan kriogenik. Pilihan bahan penanaman biasanya parafin untuk jaringan yang difiksasi formalin (FFPE) dan karboksimetilselulosa (CMC) atau gelatin untuk sampel yang dibekukan cepat, tergantung pada jenis dan kekerasan jaringan. Meskipun perannya yang menonjol dalam eksperimen histologi tradisional, penggunaan media suhu pemotongan optimal (OCT) sebagai bahan penanaman untuk MALDI-MSI telah diperingatkan sebelumnya karena kontribusinya terhadap penekanan ion, sehingga mengurangi sensitivitas deteksi analit [ 8 , 9 ]. Setelah penanaman dan pemotongan, potongan jaringan dipasang ke slide kaca menggunakan pemasangan penangas air untuk jaringan yang dipotong mikrotom dan pemasangan pencairan atau pita perekat dua sisi untuk jaringan yang dipotong kriogenik. Kaca slide indium tin oxide (ITO) direkomendasikan untuk pemasangan jaringan sehingga MSI dan mikroskop cahaya dapat dilakukan pada slide yang sama [ 10 ].
Alur kerja MALDI-MSI yang umum. Langkah-langkah utama meliputi: (1) Pengawetan jaringan melalui fiksasi formalin atau pembekuan cepat dan penanaman sampel. (2) Jaringan yang ditanam dipotong dan dipasang pada slide ITO. Langkah-langkah pemrosesan sampel seperti penghilangan lilin, pengambilan antigen, dan pencucian dapat dilakukan berdasarkan analit yang diinginkan. (3) Sampel dapat mengalami pencernaan enzimatik. Misalnya, tripsin dan PNGase F diterapkan untuk analisis peptida dan N- glikana secara berurutan. (4) Matriks diendapkan ke jaringan dengan penyemprotan atau sublimasi. (5) Data diperoleh dalam MS yang digabungkan dengan sumber MALDI. (6) Pembuatan gambar distribusi ion dengan memetakan intensitas ion yang diperoleh dari MS ke lokasi tertentu dalam jaringan, yang menciptakan peta spasial distribusi analit. Gambar dibuat menggunakan perangkat lunak BioRender.
Setelah dipasang, jaringan FFPE dideparafinisasi dan didehidrasi menggunakan rendaman gradien xilena dan etanol, diikuti dengan pengambilan antigen dan pembelahan enzimatik. Sebaliknya, jaringan beku segar menjalani pengeringan, langkah pencucian, fiksasi cepat, atau pencernaan enzimatik, yang mungkin melibatkan reagen seperti etanol dan cairan Carnoy (campuran 60% etanol, 30% kloroform, dan 10% asam asetat v/v/v) [ 11 ]. Biasanya, tripsin digunakan untuk peptida MALDI-MSI dan PNGase F untuk N -glycan MALDI-MSI [ 12 ]. Sementara jaringan FFPE dan snap frozen mampu untuk peptida dan N -glycan MALDI-MSI, sampel snap frozen memerlukan langkah pencucian tambahan untuk menghilangkan lipid karena efisiensi ionisasinya jauh lebih besar daripada peptida dan N -glycan, yang menyebabkan penekanan sinyal analit. Meskipun jaringan FFPE telah digunakan untuk analisis lipid, sampel beku cepat masih lebih disukai untuk menghindari hilangnya analit lipid selama langkah penghilangan lilin [ 13 ]. Matriks kemudian diaplikasikan ke bagian yang dipasang, sering kali menggunakan penyemprot atau sublimasi. Jika diinginkan, sublimasi adalah pilihan pertama karena teknik bebas pelarut ini meminimalkan difusi zat terlarut melintasi daerah jaringan, sehingga menghasilkan delokalisasi spasial [ 14 ]. Matriks berfungsi sebagai senyawa penting untuk mendorong desorpsi dan ionisasi analit yang diinginkan dalam MALDI-MSI [ 15 ]. Slide yang dilapisi matriks kemudian dimuat ke dalam MS, tempat laser menembakkan matriks dengan analit yang diekstraksi dari jaringan. Energi laser diserap oleh matriks dan analit dalam jaringan, sehingga memfasilitasi ionisasi. Molekul terionisasi ini kemudian dianalisis berdasarkan rasio massa terhadap muatan ( m / z ). Spektrum massa diperoleh di setiap lokasi saat laser ditembakkan ke titik berbeda di permukaan jaringan, menciptakan peta spasial intensitas ion, yang diproses menjadi gambar distribusi molekuler yang mencerminkan komposisi jaringan, menggunakan perangkat lunak analisis data dan algoritma yang kompatibel.
2 Aplikasi MALDI-MSI di Tahun 2024
MALDI-MSI semakin banyak diterapkan di berbagai bidang, mengubah cara informasi molekuler yang terpecahkan secara spasial digunakan. Bagian ini menyoroti bidang-bidang utama tempat MALDI-MSI telah memberikan dampak signifikan selama setahun terakhir.
2.1 Penemuan Biomarker dan Patologi Penyakit
MALDI-MSI diterapkan secara luas dalam onkologi karena kemampuannya untuk memberikan wawasan tentang heterogenitas tumor dan lingkungan mikro. Dengan menggunakan MALDI-MSI, para peneliti mengidentifikasi peptida kolagen matriks ekstraseluler (ECM) untuk membedakan karsinoma duktal non-invasif in situ dari kanker payudara invasif, menyoroti biomarker potensial untuk mengkarakterisasi kanker payudara [ 16 ]. Perubahan N -glycans dan protein dalam ECM juga diusulkan sebagai prediktor perkembangan kanker prostat setelah pasca prostatektomi [ 17 ]. Lebih jauh lagi, MALDI-MSI telah menjadi teknik yang banyak digunakan dalam penelitian neurologis karena kemampuannya untuk memetakan perubahan lipid, metabolit, glikana, dan protein dalam jaringan otak secara tidak bias secara spasial. Sorotan penelitian penting pada tahun 2024 meliputi identifikasi akumulasi gangliosida pada plak beta amiloid pada penyakit Alzheimer [ 18 ], identifikasi perubahan lipid yang terkait dengan skizofrenia [ 19 ] dan demonstrasi kemampuan MALDI-MSI untuk membedakan daerah lesional dari perilesional pada epilepsi menggunakan jaringan tetap pasca operasi (Gambar 3A ) [ 20 ].
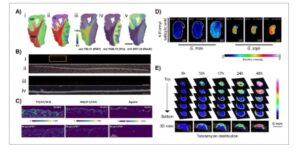
GAMBAR 3
Buka di penampil gambar
Beragam aplikasi MALDI-MSI. (A) Peptida MALDI-MSI dari penampang displasia kortikal fokal. (i, ii) Segmentasi spasial materi abu-abu (GM), materi putih (WM), perilesional (PL) dan daerah lesional (L). (iii–v) Gambar distribusi ion peptida dari protein dasar mielin (MBP, m / z 726.41), vimentin (Vim, m / z 1428.72) dan nukleus neuronal (NeuN, m / z 2407.26). Diadaptasi dan dimodifikasi dari Cagnoli et al. [ 20 ] (gbr. 3) dengan izin dari John Wiley and Sons. (B) (i) Gambar distribusi ion zolpidem ( m / z 308.17578, ditunjukkan dengan sinyal merah) dan (ii) gambar optik overlay dari sampel rambut dari individu yang menelan obat tersebut. (iii) Gambar distribusi ion zolpidem, dan (iv) gambar optik overlay rambut yang direndam. Diadaptasi dan dimodifikasi dari Ji et al. [ 22 ] (gbr. S1 dan S2) dengan izin dari Elsevier. (C) Gambar distribusi ion fosfatidilkolin (PC (32:0), m / z 734.56), sphingolipid (SM (d17: 1 / 19: 0), m / z 731.60) dan arginin ( m / z 175.11) dalam sampel kulit kontrol dan yang dirawat dengan nikel. Diadaptasi dan dimodifikasi dari Rezaei et al. [ 26 ] (gbr. 4) dengan izin dari John Wiley and Sons. (D) Gambar distribusi ion asam 4-formilsalisilat ( m / z 166.026) dalam kedelai budidaya ( G. max ) dan liar ( G. soja ). Diadaptasi dan dimodifikasi dari Yin et al. [ 39 ] (gbr. 5) dengan izin dari Elsevier. (E) Gambar distribusi ion tobramycin ( m / z 468.26) dalam koloni biofilm P. aeruginosa pada beberapa tahap pasca-perawatan dengan perspektif tiga dimensi. Diadaptasi dan dimodifikasi dari Shen et al. [ 41 ] (gbr. 6) dengan izin dari Elsevier.
2.2 Farmakologi Spasial
Investigasi pengiriman obat, penetrasi, dan akumulasi sangat penting dalam farmakokinetik, khususnya untuk mengkarakterisasi penyerapan, distribusi, metabolisme, dan ekskresi (memungkinkan pengiriman yang ditargetkan dan memahami perubahan konsentrasi temporal. Pada tahun 2024, beberapa studi MALDI-MSI dilakukan untuk tujuan ini pada berbagai jenis sampel.
Dengan menggunakan MALDI-MSI, Tenebro dan rekan-rekannya melaporkan akumulasi rotenon ( m / z 395.1495, [M + H] + ), senyawa bioaktif pada tanaman dengan sifat anti-kanker, di wilayah korteks ginjal tikus setelah 24 jam pemberian obat. Para penulis juga mencatat deteksi ion dengan m / z yang sama dalam sampel kontrol. Namun, ion ini menunjukkan pemisahan mobilitas ion yang berbeda dibandingkan dengan yang diamati pada ginjal yang diberikan obat dan standar rotenon, yang menyoroti pentingnya mobilitas ion dalam karakterisasi obat [ 21 ].
Dalam studi lain, peneliti mengamati distribusi obat hipnotik zolpidem di tengah-tengah batang rambut tunggal ketika obat tersebut ditelan. Sebaliknya, ketika rambut terkontaminasi secara sengaja, obat tersebut hanya dapat diamati di lapisan luar (Gambar 3B ) [ 22 ]. Hasil ini menunjukkan bahwa MALDI-MSI dapat menjadi alat yang berguna dalam konteks forensik untuk membedakan antara menelan obat dan potensi kontaminasi eksogen.
2.3 Aplikasi dalam Dermatologi
Penggunaan MALDI-MSI dalam model kulit merupakan area penelitian yang relatif baru tetapi berkembang pesat. Berberin, alkaloid yang berasal dari tumbuhan, terbukti dapat menembus lapisan epidermis dan dermis melalui penghantaran transdermal menggunakan susunan jarum mikro [ 23 ], yang menyiratkan kegunaan MALDI-MSI dalam mengevaluasi penetrasi obat, tetapi menggunakan model kulit ex vivo. Studi lain meneliti akumulasi penghambat protein kinase, selumetinib dan produk degradasinya pada apendiks kulit, yang dapat memberikan wawasan dalam strategi formulasi obat untuk kemanjuran yang diperpanjang [ 24 ]. MALDI-MSI juga digunakan untuk menyelidiki apakah benzalkonium klorida, antiseptik yang digunakan dalam produk antimikroba dapat menembus kulit manusia [ 25 ].
Selain menganalisis senyawa yang ditargetkan, MALDI-MSI juga dapat digunakan sebagai pendekatan omik spasial untuk menyelidiki perubahan biologis global. Misalnya, para peneliti telah mengidentifikasi regulasi lipid atau metabolit yang berbeda sebagai respons terhadap paparan nikel (Gambar 3C ) [ 26 ], atau pada jaringan parut hipertrofik [ 27 ], dibandingkan dengan jaringan normal. Temuan ini dapat memandu penelitian lebih lanjut yang bertujuan untuk mengembangkan perawatan atau pengobatan yang relevan.
2.4 Metabolisme Tumbuhan
MALDI-MSI juga telah diadaptasi secara luas dalam penelitian tanaman, mulai dari tujuan sederhana untuk memvisualisasikan akumulasi senyawa bioaktif atau metabolit [ 28 – 30 ] hingga memahami fisiologi pertumbuhan, perkembangan, atau respons tanaman terhadap stres [ 31 – 33 ]. Alur kerja MALDI-MSI pada tanaman sering kali rumit karena integritas jaringan yang buruk [ 34 ], tetapi solusi inovatif baru-baru ini telah muncul untuk mengatasi tantangan ini. salah satu solusi tersebut adalah teknik planarisasi jaringan beku berbantuan medan elektromagnetik baru, yang dirancang untuk mengatasi morfologi jaringan tanaman yang tidak teratur [ 35 ]. Yang menarik, pendekatan baru yang diperkenalkan tahun lalu, disebut RhizoMAP, memungkinkan untuk mempelajari interaksi antara akar tanaman dan tanah di sekitarnya di rizosfer tanpa mengganggu organisasi akar-tanah [ 36 ].
Salah satu terobosan MALDI-MSI paling signifikan pada tahun 2024 adalah penggunaan MALDI-2 (post ionisation) dalam penelitian tanaman. Dalam penelitian ini, penulis mengungkapkan lokalisasi metabolit bioaktif yang berbeda di berbagai kompartemen daun, seperti mesofil, epidermis, dan lapisan berkas pembuluh [ 37 ]. Memahami distribusi senyawa bioaktif dalam tanaman kemudian dapat diterjemahkan ke dalam penelitian medis. Kemampuan translasi ini lebih lanjut disorot dalam sebuah penelitian yang diterbitkan pada tahun yang sama, di mana etil caffeate dan homoorientin ditemukan terlokalisasi di eksokarp akar ginseng primer [ 38 ]. Kedua senyawa tersebut diuji untuk efek pro-inflamasinya secara in vitro dalam penelitian yang sama, yang menunjukkan bahwa penghilangan eksokarp kuning kecoklatan dapat mengurangi efek samping setelah konsumsi ginseng [ 38 ].
MALDI-MSI juga berguna dalam menguraikan mekanisme molekuler yang terkait dengan pertahanan tanaman, respons stres atau pertumbuhan dan perkembangan. Dalam upaya untuk mengkarakterisasi kedelai liar versus yang dibudidayakan, para peneliti mengungkap penurunan regulasi metabolit yang terkait dengan toleransi stres pada benih yang dibudidayakan (Gambar 3D ) [ 39 ]. Temuan-temuan ini menunjukkan hilangnya kemampuan beradaptasi lingkungan pada kedelai selama proses domestikasi, oleh karena itu persilangan dengan kedelai liar dapat membangun kembali sifat ekologi yang lebih kuat pada varietas yang dibudidayakan. Deteksi fitohormon menggunakan MALDI-MSI juga memungkinkan penyelidikan regulasi pertumbuhan tanaman selama absisi kelopak pada pir harum Korla, suatu proses yang penting dalam menentukan kualitas buah [ 40 ].
2.5 Aplikasi pada Organisme Lain
Selain pada hewan dan tumbuhan, MALDI-MSI telah diterapkan di bidang kingdom lain selama dekade terakhir. Tahun 2024 adalah saat kita menyaksikan upaya para peneliti untuk mendorong batasan lebih jauh di area ini.
Yang paling menonjol, alur kerja non-embedding moisture-assisted cryo-section (MACS) ditetapkan untuk metabolit tiga dimensi (3D) MALDI-MSI dari koloni mikroorganisme dalam biofilm [ 41 ]. Metode ini diuji dalam biofilm Pseudomonas aeruginosa dan Staphylococcus aureus , yang menggarisbawahi keserbagunaannya. Distribusi antibiotik tobramisin dilacak dalam koloni biofilm, yang mengungkapkan pola lokalisasi non-difusif di lapisan luar (Gambar 3E ). Disarankan bahwa karena tobramisin memerlukan rantai transpor elektron aktif untuk diangkut ke dalam bakteri, sel-sel dalam biofilm menunjukkan resistensi intrinsik karena dormansi [ 41 ].
Langkah persiapan sampel baru juga diperkenalkan untuk mengatasi keterbatasan pada pencitraan koloni bakteri dengan permukaan heterogen [ 42 ]. Alur kerja ini melibatkan kultur mikroorganisme secara tidak langsung pada media agar, yang memiliki membran polimer di antaranya. Membran ini kemudian dapat dihilangkan bersama dengan koloni, meninggalkan metabolit bakteri yang telah dilepaskan melalui membran ke agar. MALDI-MSI dapat dilakukan pada media agar yang dipotong dengan permukaan datar, sehingga meningkatkan sensitivitas dan kualitas gambar [ 42 ]. Studi MALDI-MSI menarik lainnya termasuk membandingkan kelimpahan metabolit pada Trichodesmium cyanobacterium liar dan yang dibudidayakan [ 43 ], mengidentifikasi perubahan glikosfingolipid selama pematangan parasit cacing hati ( Fasciola hepatica ) [ 44 ] dan melacak dinoflagellata Symbiodiniaceae di karang pembentuk terumbu [ 45 ].
3 Kemajuan Terbaru: Menuju Multi-Omik Spasial pada Resolusi Sel Tunggal
MALDI-MSI adalah teknologi baru yang telah mengalami kemajuan signifikan selama bertahun-tahun, sebagaimana ditegaskan oleh jumlah publikasi terkait yang terus meningkat. Meskipun demikian, masih ada tantangan dan keterbatasan yang terkait dengan teknologi ini. Para peneliti berusaha keras untuk mendeteksi sebanyak mungkin analit yang diinginkan dengan sensitivitas tinggi dan konten informasi yang kaya sambil mencapai resolusi spasial tertinggi. Tidak seperti LC-MS/MS, akuisisi data MALDI-MSI yang tidak ditargetkan pada umumnya tidak melibatkan MS/MS, yang memecah analit untuk pemetaan struktural guna memperoleh identifikasi yang meyakinkan. Akibatnya, memperoleh spektrum MS yang kompleks dan informatif sangat penting dalam eksperimen MALDI-MSI mandiri. Namun, MALDI-MSI sering kali mengalami sensitivitas rendah pada resolusi spasial tinggi karena jumlah analit yang dihilangkan di area pengambilan sampel kecil oleh laser menentukan sensitivitasnya. Di bagian ini, kami menyoroti beberapa kemajuan besar pada tahun 2024 yang bertujuan untuk meningkatkan eksperimen MALDI-MSI.
3.1 Persiapan Sampel
Persiapan sampel merupakan langkah penting dalam eksperimen MALDI-MSI, yang pada akhirnya memengaruhi hasil, sensitivitas, resolusi, dan kualitas gambar. Oleh karena itu, teknik inovatif terus dikembangkan dalam tahap awal alur kerja MALDI-MSI ini.
3.1.1 Derivatisasi Pada Slide
Derivatisasi meningkatkan stabilitas dan kemampuan deteksi analit, khususnya N -glikans. Namun, derivatisasi pada jaringan selalu menjadi tantangan bagi analisis MSI karena ketidakmampuan untuk melakukan pembersihan sampel. Kehadiran molekul lain pada jaringan dapat menyebabkan persaingan untuk ionisasi, yang menyebabkan penekanan ionisasi dan berkurangnya sensitivitas untuk analit tertentu.
Pada tahun 2024, studi memperkenalkan pendekatan baru untuk derivatisasi pada jaringan menggunakan jaringan FFPE yang kompatibel dengan analisis MSI. Misalnya, Cumin et al. [ 46 ] menyempurnakan protokol sebelumnya dengan menggunakan PyAOP (7-Azabenzotriazol-1-yloxy)tripyrrolidinophosphonium hexafluorophosphate) dan metilamina sebagai reaktan untuk amidasi stabil glikana sialilasi [ 47 , 48 ]. Studi mereka menunjukkan peningkatan intensitas spesies sialilasi pada massa yang lebih tinggi, sementara intensitas m / z 1663.5 (Hex5HexNAc4) menurun setelah derivatisasi menggunakan metode 15 menit mereka. Metode ini juga kompatibel dengan resolusi hampir sel tunggal, memberikan metode yang kuat dan andal untuk derivatisasi N -glycan dalam MALDI MSI [ 46 ]. Hasilnya menunjukkan bahwa intensitas puncak N- glikana netral dapat dipengaruhi oleh hilangnya spesies tersialilasi, yang menekankan pentingnya derivatisasi dalam eksperimen MSI yang melibatkan N- glikana.
3.1.2 Aplikasi Matriks
Pemilihan matriks sangat penting untuk keberhasilan percobaan MALDI-MSI, karena mempengaruhi pencocokan polaritas antara matriks dan analit, pilihan pelarut, stabilitas kimia selama pencitraan jangka panjang dan interferensi latar belakang. Matriks yang paling umum digunakan meliputi CHCA (α-Cyano-4-hydroxycinnamic acid), DHB (2,5-dihydroxybenzoic acid), DHAP (2,6-Dihydroxyacetophenone) untuk glikopeptida dan fosfopeptida, atau 9-AA (9-aminoacridine), SA (sinapinic acid) dan DAN (1,5-diaminonaphthelene) untuk analisis MSI lipid, peptida, glycan dan protein. Namun, matriks ini tidak cocok untuk MSI metabolit, karena interferensi dari ion matriks, yang menekan deteksi analit dengan berat molekul rendah [ 49 ].
Pada tahun 2024, matriks baru telah diperkenalkan untuk meningkatkan sensitivitas untuk analit yang berbeda. Bao et al. [ 49 ] menunjukkan penggunaan 2-hidroksi-5-nitro-3-(trifluorometil)piridina (HNTP) sebagai matriks untuk MSI metabolit dalam rentang m / z 60–1500 yang berhasil mendeteksi 152 metabolit dalam jaringan otak tikus pada resolusi 150 µm. Matriks ini mendeteksi 31 analit lebih banyak daripada DHB [ 49 ]. Meskipun resolusi spasial tidak pada tingkat sel tunggal, tidak ada masalah dengan delokalisasi atau resolusi yang lebih rendah yang dilaporkan. Selain itu, Chen et al. [ 50 ] menguji asam 2,4-dihidroksi-5-nitrobenzoat (DHNBA), yang mengungguli DHB dalam mendeteksi fitohormon dalam jaringan tanaman [ 51 , 52 ]. Lima sitokinin isoprenoid (tZ, DHZ, ABA, IAA dan ACC) divisualisasikan dengan MALDI-MSI untuk pertama kalinya, menyoroti pentingnya pemilihan matriks dalam meningkatkan deteksi analit [ 50 ]. Lebih lanjut, Liang et al. [ 53 ] menunjukkan bahwa derivatif nitro indole berfungsi sebagai matriks superior dalam MALDI-MSI, menunjukkan sensitivitas yang lebih tinggi dibandingkan dengan matriks yang digunakan secara rutin (DHB, CHCA, SA, DAN dan 9-AA) untuk lipid, peptida, protein, glikana dan PFOS MSI dalam mode ionisasi positif dan negatif [ 53 ]. Temuan ini menyoroti pentingnya kritis pemilihan matriks dalam meningkatkan sensitivitas dan akurasi MALDI-MSI.
Ukuran kristal memainkan peran penting dalam resolusi spasial, menjadikannya pertimbangan penting dalam eksperimen MALDI-MSI. Pada tahun 2024, kemajuan signifikan dalam metode kristalisasi dan deposisi matriks berkontribusi pada peningkatan resolusi. Teknik berbasis semprotan yang melibatkan penggunaan pelarut organik biasanya menghasilkan kristal berukuran lebih besar dari 10 µm [ 54 ]. Sebagai alternatif, metode berbasis sublimasi telah dikembangkan di mana matriks fase padat berubah menjadi gas di bawah panas dan vakum, yang kemudian melapisi slide yang didinginkan di bawah ruang dingin [ 54 ]. Metode ini pertama kali berhasil diterapkan pada studi MALDI-MSI pada tahun 2007 [ 14 ]. Metode berbasis sublimasi menciptakan kristal yang lebih halus dan mencegah analit dari pelarutan ulang dan migrasi lateral [ 54 ].
Penelitian pada tahun 2024 menjajaki cara untuk mengoptimalkan kinerja matriks, seperti menyempurnakan suhu kristalisasi untuk mengurangi ukuran kristal [ 54 ]. Selain itu, penggunaan jangkar polimer konjugat telah diteliti untuk meminimalkan penguapan matriks dalam vakum tinggi [ 55 ]. Pencapaian penting pada tahun 2024 adalah pengembangan metode yang mencapai resolusi 1,5-µm untuk MSI sel tunggal dengan menggunakan sistem penyemprotan pneumatik ultra-halus dalam mikroprobe pemindaian tekanan atmosfer MALDI-MSI (AP-SMALDI MSI) [ 56 ]. Kemajuan ini menjanjikan peningkatan lebih lanjut dalam resolusi spasial dalam eksperimen MALDI-MSI.
3.1.3 Pendekatan Ekspansi Jaringan
Mikroskopi ekspansi pertama kali ditemukan pada tahun 2015 oleh Chen, Tillberg dan Boyden untuk mikroskopi resolusi super dengan mikroskop terbatas difraksi dengan memperluas dan memperbesar sel dan jaringan tetap secara fisik menggunakan gel polielektrolit yang dapat membengkak [ 57 ]. Pada tahun 2024, protokol ekspansi jaringan yang kompatibel dengan MALDI-MSI telah diselidiki. Selama mikroskopi ekspansi, sampel diperluas secara isotropik, memungkinkan pengamatan mikrostruktur jaringan yang lebih rinci. Untuk memungkinkan adaptasi MALDI-MSI, protokol telah dimodifikasi untuk memenuhi pencegahan kriogenik dan penyusutan dalam kondisi vakum menggunakan polisakarida natrium hialuronat sebagai dukungan struktural. Ekspansi dua kali lipat dicapai melalui teknik ini untuk meningkatkan detail morfologi [ 58 ], yang selanjutnya menoleransi akuisisi data MALDI-MSI pada tingkat sel tunggal atau bahkan subseluler.
3.2 Multi-Omik Spasial
Multiomik spasial melibatkan ko-lokalisasi tepat informasi yang terpecahkan secara spasial di beberapa lapisan molekuler, seperti transkriptom, proteom, glikom, dan metabolom, yang memberikan wawasan holistik ke dalam sistem biologis kompleks.
3.2.1 Penggabungan MALDI-MSI dengan Mikrodiseksi Penangkapan Laser
Laser capture microdissection (LCM) memungkinkan peneliti untuk secara selektif mengangkat daerah jaringan untuk analisis hilir. Daerah yang diinginkan ini sering didefinisikan menggunakan pewarnaan imunohistokimia, yang hanya dapat mendeteksi beberapa analit yang ditargetkan karena terbatasnya ketersediaan antibodi, sehingga membatasi kemampuan multipleks. Di sisi lain, MALDI-MSI menawarkan pandangan komprehensif dari lanskap molekuler melalui pemetaan daerah yang diinginkan yang tidak bias. Namun, slide ITO yang umum digunakan dalam MALDI-MSI tidak kompatibel dengan LCM, yang memerlukan slide membran polietilen naftalat (PEN). Akibatnya, analisis ini harus dilakukan pada bagian jaringan yang terpisah daripada bagian tunggal. Sementara pendekatan ini dapat diterima untuk sebagian besar jenis sampel, pendekatan ini menghadirkan tantangan yang signifikan untuk jaringan seperti paru-paru yang mengembang, di mana ruang berisi udara dan struktur 3D yang tidak teratur dapat menyebabkan perbedaan substansial bahkan antara bagian yang berurutan [ 12 ]. Keterbatasan ini membuat alur kerja tidak cocok untuk multiomik spasial sel tunggal, di mana bagian yang berurutan tidak dapat diperoleh.
Pada tahun 2021, Mezger et al. [ 59 ] mencoba melakukan LCM langsung pada slide ITO, tetapi pendekatan tersebut terbukti kurang menguntungkan untuk identifikasi protein [ 59 ]. Untuk mengatasi keterbatasan ini, Truong et al. mengevaluasi kelayakan penggunaan slide membran PEN untuk MALDI-MSI sebagai pengganti slide konduktif ITO tradisional, karena slide non-konduktif kompatibel dengan sistem timsTOF fleX (Bruker Daltonics, Jerman) [ 60 ]. Hasil penelitian mereka menunjukkan bahwa intensitas relatif analit sebanding untuk kedua jenis slide. Selain itu, pengelompokan hierarkis data MS yang diselesaikan secara spasial menghasilkan peta segmentasi yang hampir identik terlepas dari jenis slide dan selaras dengan informasi histologi pada bagian yang diwarnai H&E berturut-turut. Para penulis juga mengusulkan pendekatan multiomik spasial yang inovatif, di mana MALDI-MSI lipidomik pertama kali digunakan untuk menentukan daerah yang diminati untuk pemetaan LCM, diikuti oleh proteomik LC-MS/MS, yang memungkinkan korelasi yang lebih baik antara dua set data omik. Data MSI, atau lanskap Lab SCiLS, kemudian diekspor ke perangkat lunak pemeta wilayah Bruker untuk pemetaan daerah yang diminati secara tepat. Informasi tersebut kemudian ditransfer ke sistem koordinasi mikroskop LCM untuk LCM yang tepat [ 60 ]. Dengan peningkatan kemampuan LCM untuk mengisolasi sel tunggal dan bahkan organel, pendekatan ini menandai langkah maju yang signifikan menuju multiomik spasial subseluler.
Kemajuan teknologi terkini telah bergeser secara signifikan dari analisis jaringan massal tradisional ke kemampuan untuk memeriksa heterogenitas seluler di seluruh jaringan, menawarkan pandangan komprehensif tentang fitur molekuler pada tingkat tipe sel individual [ 61 ]. Kemajuan ini dicontohkan oleh pengembangan Deep Visual Proteomics (DVP), pendekatan perintis yang dicetuskan oleh Mund et al. [ 62 ] pada tahun 2022, yang memungkinkan analisis proteom tipe sel tunggal spasial. Dengan menggabungkan pencitraan resolusi tinggi, fenotipe sel tunggal berbasis AI, mikrodiseksi laser otomatis, dan MS ultrasensitif, DVP menyediakan alat yang ampuh untuk membedah lanskap proteomik jaringan pada tingkat detail yang belum pernah terjadi sebelumnya [ 63 ]. Mengingat kemampuan DVP untuk memberikan profil proteomik terperinci dari tipe sel individual, pada tahun 2024, Zheng et al. [ 64 ] menunjukkan keakuratan dan kekokohannya dalam kasus gabungan limfoma Hodgkin klasik (cHL) dan limfoma limfositik kecil (SLL) pada satu pasien. Temuan mereka mengungkap profil proteom yang berbeda pada populasi cHL dan SLL, yang menyoroti ketidakterkaitan klonal mereka, dan menyarankan bahwa strategi seperti kemoterapi standar dan penghambatan interleukin-4 dapat mengatasi resistensi kemoterapi, yang menawarkan alternatif untuk transplantasi sumsum tulang. Wawasan spesifik jenis sel dari DVP ini dapat memandu perawatan onkologis yang dipersonalisasi [ 64 ].
3.2.2 Penggabungan MALDI-MSI dengan Imunohistokimia
Baru-baru ini, Bindi et al. [ 65 ] mengeksplorasi potensi teknik yang baru dikembangkan yang disebut MALDI-HiPLEX-IHC, yang menggabungkan kekuatan multiplexing MSI dengan imunohistokimia. Metode ini melibatkan pewarnaan jaringan dengan berbagai antibodi yang dikaitkan dengan tag massa yang dapat difotokleavabel, yang memungkinkan pencitraan biomolekul yang sangat multipleks dan tertarget dalam jaringan. Dalam studi bukti konsep, penulis menunjukkan alur kerja yang secara berurutan mengintegrasikan MALDI-HiPLEX-IHC dengan proteomik spasial yang tidak ditargetkan pada satu bagian jaringan FFPE, menggunakan karsinoma sel ginjal sel jernih klinis sebagai model. Pendekatan ini memungkinkan stratifikasi inti tumor yang secara histologis serupa dengan tingkat yang sama, berdasarkan perbedaan populasi limfosit, khususnya sel pengatur T. Lebih jauh lagi, ketika dikombinasikan dengan proteomik spasial yang tidak ditargetkan, studi ini mengungkapkan perubahan proteomik yang terkait dengan pola infiltrasi limfosit ini. Temuan ini menyoroti potensi alur kerja terpadu ini untuk memetakan dan mengkarakterisasi lingkungan molekuler limfosit yang menyusup ke tumor, memberikan wawasan berharga tentang dampak molekuler sel imun dalam lingkungan mikro tumor [ 65 ].
3.2.3 Algoritma untuk Integrasi Data Multi-Omics
Dengan munculnya tren ‘multi-omik spasial’, tantangan utama terletak pada pengintegrasian kumpulan data omik yang berbeda, tidak hanya pada tingkat spasial tetapi juga secara bioinformatika. Data yang dihasilkan dari berbagai teknik atau instrumen sering kali hadir dalam format file yang berbeda dan memerlukan perangkat lunak dan algoritma yang berbeda untuk analisis. Fusi gambar tradisional telah dipraktikkan selama lebih dari satu dekade, yang melibatkan pendaftaran bersama gambar histologi dan imunohistokimia dengan kumpulan data MALDI-MSI resolusi spasial rendah menggunakan model matematika [ 66 ]. Proses ini bergantung pada penajaman pencitraan dan algoritma prediksi model untuk mengkorelasikan wilayah yang diinginkan dalam gambar mikroskopis dan peta intensitas ion dari MALDI-MSI, biasanya melalui analisis yang diawasi dan padat karya [ 66 ]. Saat kita maju menuju resolusi sel tunggal, algoritma multi-moda berkembang untuk mengakomodasi skala ini [ 67 ] dan aplikasi pada analisis multi-omik.
Kemajuan terbaru pada tahun 2024 telah menyoroti integrasi MSI metabolomik dengan analisis transkriptomik spasial, seperti yang ditunjukkan oleh Vicari et al. [ 68 ] Pendekatan ini menggunakan aplikasi interaktif yang memanfaatkan pewarnaan histologi dan pemetaan intensitas analit untuk mengorelasikan data spasial dan molekuler secara efektif. Lebih jauh memajukan bidang ini, Schwenzfier et al. [ 69 ] memperkenalkan Fluorescence Integrated Single-Cell Analysis Script (FISCAS) yang secara otomatis menghasilkan spektrum massa sel tunggal dengan parameter morfometrik yang dioptimalkan, meningkatkan deteksi heterogenitas seluler dengan menggabungkan pewarnaan fluoresensi sampel sebelum akuisisi MALDI-MSI [ 69 ]. FISCAS memfasilitasi pemilihan otomatis wilayah pengukuran yang ditentukan secara ketat, sehingga meminimalkan akuisisi piksel di luar target. Selain itu, ia memanfaatkan algoritma yang mapan untuk segmentasi sel dan registrasi bersama, memfasilitasi kompilasi cepat spektrum sel tunggal. Dengan memungkinkan pengukuran yang lebih tepat pada resolusi sub-seluler, FISCAS secara signifikan meningkatkan potensi integrasi data multi-moda hilir, menjadikannya alat yang berharga untuk alur kerja analitis tingkat lanjut.
3.3 Instrumentasi MS, Akuisisi dan Analisis Data
Dalam beberapa tahun terakhir, instrumentasi MS untuk MALDI-MSI telah mengalami terobosan dalam resolusi spasial dan sensitivitas, serta kemampuan untuk memetakan ion fragmen pada tingkat MS/MS. Keterbatasan MALDI-MSI, seperti kurangnya data dan dimensi MS/MS untuk identifikasi yang akurat, telah diatasi secara bertahap melalui beberapa teknologi: (1) MS mobilitas ion, yang menambahkan tingkat lain dari nilai pemisahan dengan penampang tumbukan (CCS) berdasarkan konformasi ion, (2) iprm-PASEF yang memungkinkan pemetaan paralel ion fragmen pada tingkat MS/MS dan (3) MALDI-2, teknik pasca-ionisasi, meningkatkan efisiensi ionisasi. Lebih jauh lagi, algoritma analisis data juga telah ditingkatkan melalui integrasi pembelajaran mendalam untuk pemetaan presisi analit pada tingkat sel tunggal.
3.3.1 Mobilitas Ion
Mobilitas ion ( K ) diukur dan dilaporkan sebagai mobilitas ion tereduksi terbalik (1/ K 0 ), yang ditentukan oleh nilai CCS, berdasarkan bentuk ion prekursor dalam fase gas. Dalam beberapa tahun terakhir, teknologi spektrometri massa mobilitas ion terperangkap (TIMS) telah membuat kemajuan untuk memungkinkan dimensi pemisahan ion ekstra untuk memungkinkan identifikasi analit yang tepat, yang memungkinkan pemisahan analit isobarik dan isomerik (Gambar 4 ) [ 70 ]. Teknologi ini telah banyak digunakan untuk proteomik LC-MS/MS [ 71 , 72 ]. Sistem sel TIMS ganda dari instrumen timsTOF fleX (Bruker Daltonics, Jerman) memungkinkan algoritma akuisisi fragmentasi serial akumulasi paralel (PASEF) dengan menyinkronkan penyimpanan dan pelepasan ion menggunakan perangkat perangkap ion. Ini diikuti oleh pemilihan ion berbasis kuadrupol dan fragmentasi untuk memaksimalkan penggunaan ion dan meningkatkan sensitivitas untuk deteksi ion fragmen [ 73 ]. Dengan menggunakan algoritma terbaru, yang dikenal sebagai iprm-PASEF (Bruker Daltonics, Jerman), Li et al. telah menunjukkan peningkatan identifikasi peptida dalam MALDI-MSI dengan memilih hingga lima ion prekursor dengan 1/ K 0 dan rentang m / z yang telah ditentukan sebelumnya , sebagaimana didukung oleh hasil pencarian Mascot yang berhasil, tanpa mengorbankan sensitivitas dan kualitas spektrum [ 73 ]. Distribusi ion fragmen kemudian dapat divisualisasikan menggunakan versi terbaru SCiLS Lab (Bruker Daltonics, Jerman). Sementara iprm-PASEF hanya dapat dilakukan pada lima prekursor, ini masih merupakan langkah maju yang signifikan darieksperimen MALDI-MSI konvensional yang hanya menyediakan informasi MS1.
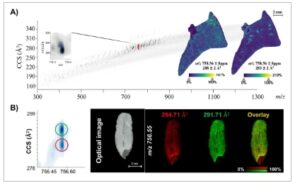
GAMBAR 4
Buka di penampil gambar
MALDI-MSI yang digabungkan dengan pemisahan mobilitas ion terperangkap (TIMS) memungkinkan diferensiasi molekul isobarik dengan pola distribusi yang berbeda. (A) Identifikasi spesies isobarik ( m / z 758.56) dengan nilai penampang tumbukan (CCS) yang berbeda pada jaringan paru-paru tikus. Gambar distribusi ion mengungkapkan pola lokalisasi spesies isobarik yang berbeda. Diadaptasi dari Ngai et al. [ 12 ] (gbr. 3) dengan izin dari John Wiley and Sons. (B) Pengamatan serupa dilakukan pada jaringan biji gandum untuk m / z 756.55, dengan satu spesies terdistribusi di seluruh biji (merah), sementara yang lain terlokalisasi di wilayah endosperma putih (hijau). Diadaptasi dan dimodifikasi dari Lau et al. [ 81 ] (gbr. 6) dengan izin dari Elsevier.
3.3.2 MALDI-2
Oversampling telah menjadi teknik untuk meningkatkan resolusi spasial dengan menggerakkan panggung dalam peningkatan yang lebih kecil daripada sinar laser. Dengan hanya satu bagian dari sinar laser yang digunakan untuk ionisasi, ukuran titik yang lebih kecil dihasilkan [ 74 ]. Namun, kompromi dalam sensitivitas dapat terjadi [ 75 ]. Untuk mengatasi hal ini, teknik pasca-ionisasi laser MALDI-2, menunjukkan jumlah analit ganda yang dideteksi per piksel, berdasarkan brosur produk timsTOF fleX MALDI-2 (Bruker Daltonics, Jerman). Akurasi posisi panggung memainkan bagian penting dalam teknik ini, karena ukuran langkah minimum dan reproduktifitas sangat penting untuk menghindari stripping dan artefak lainnya [ 76 ]. MicroGRID oleh Bruker Daltonics (Bruker Daltonics, Jerman) dapat mengatasi hal ini untuk memungkinkan eksperimen MSI sel tunggal dan subseluler untuk pergerakan panggung yang tepat. juga telah menunjukkan teknik pasca-ionisasi menggunakan spektrometer massa Orbitrap Elite (Thermo Fisher Scientific GmbH, Jerman) yang digabungkan dengan sumber MALDI/ESI tekanan menengah (Spectroglyph LLC, AS) dengan laser Nd:YAG (Nano L-DPSS) (Litron Lasers, Inggris) sebagai MALDI-2, menghasilkan resolusi spasial tinggi sebesar 6 µm ukuran piksel [ 76 ].
3.4 Pembelajaran Mesin dan Pembelajaran Mendalam
Analisis dan interpretasi MALDI-MSI dari data molekuler yang kompleks sekarang dapat ditingkatkan dengan integrasi teknik pembelajaran mesin (ML) dan pembelajaran mendalam (DL). Model ML yang dilatih menggunakan data MALDI-MSI telah menunjukkan hasil diagnostik yang akurat. Misalnya, sebuah studi tentang karsinoma sel skuamosa kulit menggunakan ML yang diawasi untuk mencapai akurasi prediktif 92,3%, dengan validasi patologis melebihi 99% [ 77 ]. Demikian pula, sebuah studi tentang karsinoma sel basal mencapai akurasi klasifikasi 99%, dengan profil metabolom yang mengidentifikasi 189 sinyal signifikan sebagai penanda tumor potensial [ 78 ]. Contoh-contoh ini menggambarkan potensi MALDI-MSI yang ditingkatkan ML dalam diagnosis penyakit klinis.
Beberapa platform telah mengintegrasikan ML dan DL ke dalam alur kerja analisis data MALDI-MSI pada tahun 2024. Misalnya, METASPACE-ML, penerus platform METASPACE yang ada untuk anotasi metabolit, diperkenalkan. Model ML ini dilatih pada kumpulan data hewan dan tumbuhan yang luas, dan terbukti mengungguli pendekatan berbasis aturan konvensional, memberikan presisi yang lebih tinggi pada ambang batas tingkat penemuan palsu (FDR) yang rendah dan kemampuan yang lebih besar untuk mendeteksi jejak metabolit [ 79 ]. Lebih jauh lagi, MEISTER, yang menggunakan teknik pembelajaran mendalam untuk meningkatkan pemrosesan data dan throughput untuk analisis jaringan dan sel tunggal, dikembangkan. Kerangka kerja MS canggih ini menggabungkan inovasi seperti: (i) metode rekonstruksi sinyal pembelajaran mendalam untuk meningkatkan resolusi massa, (ii) teknik registrasi gambar multimoda untuk rekonstruksi jaringan 3D dan analisis kimia kuantitatif berikutnya dan (iii) pendekatan komputasi untuk mengintegrasikan profil kimia spesifik sel dengan data pencitraan jaringan [ 80 ]. Dengan MEISTER, para peneliti telah membangun perpustakaan kimia khusus jenis sel dari lebih dari 13.000 sel tunggal di berbagai wilayah otak tikus. Perpustakaan tersebut kemudian dipetakan ke gambar jaringan, yang menunjukkan heterogenitas dalam distribusi lipid pada tingkat jaringan dan sel tunggal [ 80 ]. Teknik ini merupakan tonggak penting dalam integrasi omik spasial untuk karakterisasi biokimia multiskala.
4 Ringkasan dan Prospek
Selama bertahun-tahun, MALDI-MSI beralih dari teknik pencitraan molekuler baru yang digunakan untuk penelitian biomedis menjadi alat mapan dengan aplikasi yang terus berkembang. Di sini, kami menggambarkan beragam aplikasi MALDI-MSI dalam mempelajari berbagai jenis organisme. Sementara MALDI-MSI terus memainkan peran penting dalam studi pengembangan penyakit dan obat, kami meramalkan pertumbuhan aplikasi MALDI-MSI ke bidang yang lebih beragam. Misalnya, perdebatan yang berkembang seputar polusi zat perfluoroalkil dan polifluoroalkil (PFAS) telah membuat para peneliti tertarik untuk menyelidiki akumulasi mikroplastik dalam jaringan mamalia atau tumbuhan. MALDI-MSI juga telah menunjukkan tujuan keanekaragaman hayati dan pelestarian alam, dengan kemampuan untuk mengkarakterisasi interaksi metabolik dalam ekosistem. MALDI-MSI juga dapat menjadi teknik yang berharga dalam industri makanan dan kosmetik, terutama untuk pengujian alergen.
Kemajuan signifikan dalam MALDI-MSI terlihat pada tahun 2024, khususnya dalam teknik persiapan sampel, instrumentasi untuk meningkatkan resolusi spasial dan pemisahan mobilitas ion, integrasi multiomik spasial, dan perluasan ke analisis sel tunggal. Integrasi kecerdasan buatan juga dapat mendorong MALDI-MSI maju, meningkatkan analisis data, mengotomatiskan alur kerja, dan menjadi lebih ramah pengguna. Dengan evolusinya yang berkelanjutan menjadi alat multidisiplin, masa depan MALDI-MSI tampak menjanjikan.

